
comparacion de listas
Publicado por alvaro (1 intervención) el 07/11/2017 13:29:33
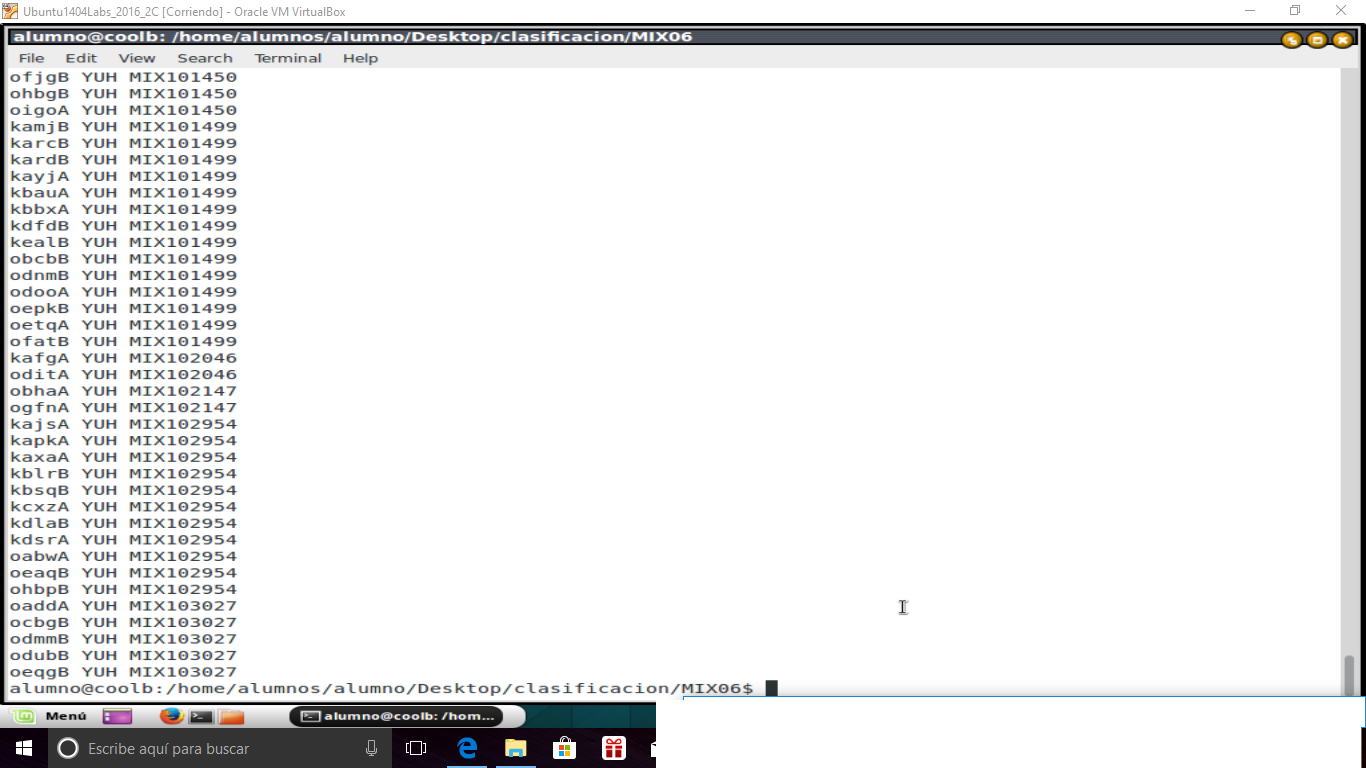
Buenas, tengo un archivo de 2914 filas y 3 columnas. La primera columna es la que me interesa: es simplemente una cadena de 5 letras.
Por otro lado tengo otro directorio con 5100 archivos '.mat':
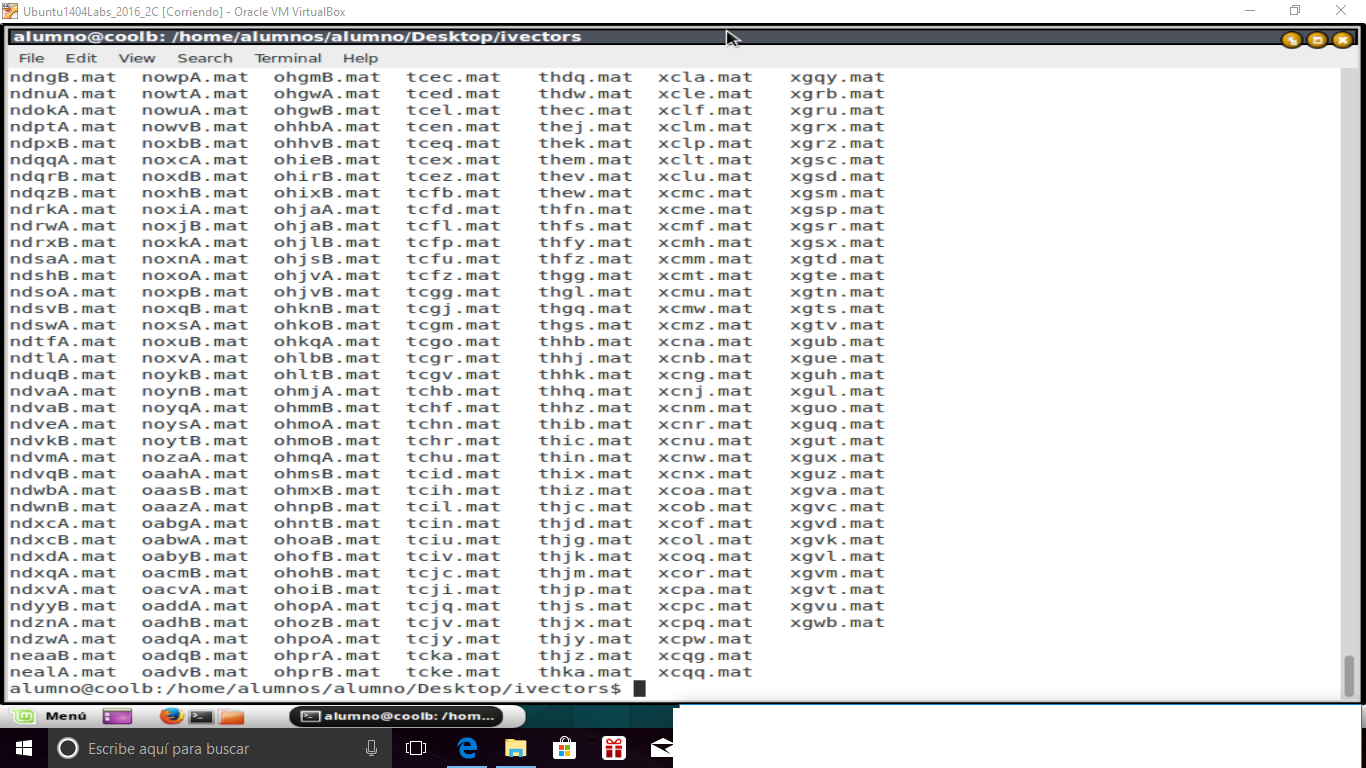
El nombre de cada archivo '.mat' coincide con la primera columna del otro directorio.
El caso es que hay nombres de archivos de la primera columna del primer directorio, que no están en los 5100 archivos '.mat' (es decir, faltan aún más archivos .mat) del otro directorio, y me gustaría saber cuales son los que NO están (para almacenarlos en una lista) sin tener que ir de 1 en 1 porque sería casi infinito XD.
Os agradecería mucho vuestra ayuda, ¿Cómo puedo hacer esto?.
Muchas gracias!!!!!
Valora esta pregunta


0
