Extraer tablas de un archivo txt
Publicado por Claudia (3 intervenciones) el 16/10/2019 21:38:38
Hola a todos!!! por favor necesito ayuda con algún código que me permita extraer una tabla de un archivo txt que contiene mas tablas y frases.
como lo puedo hacer?
Les adjunto una foto para que vean lo que quiero decir. enmarcada en rojo está la tabla que necesito copiar y pasarla a otro archivo.
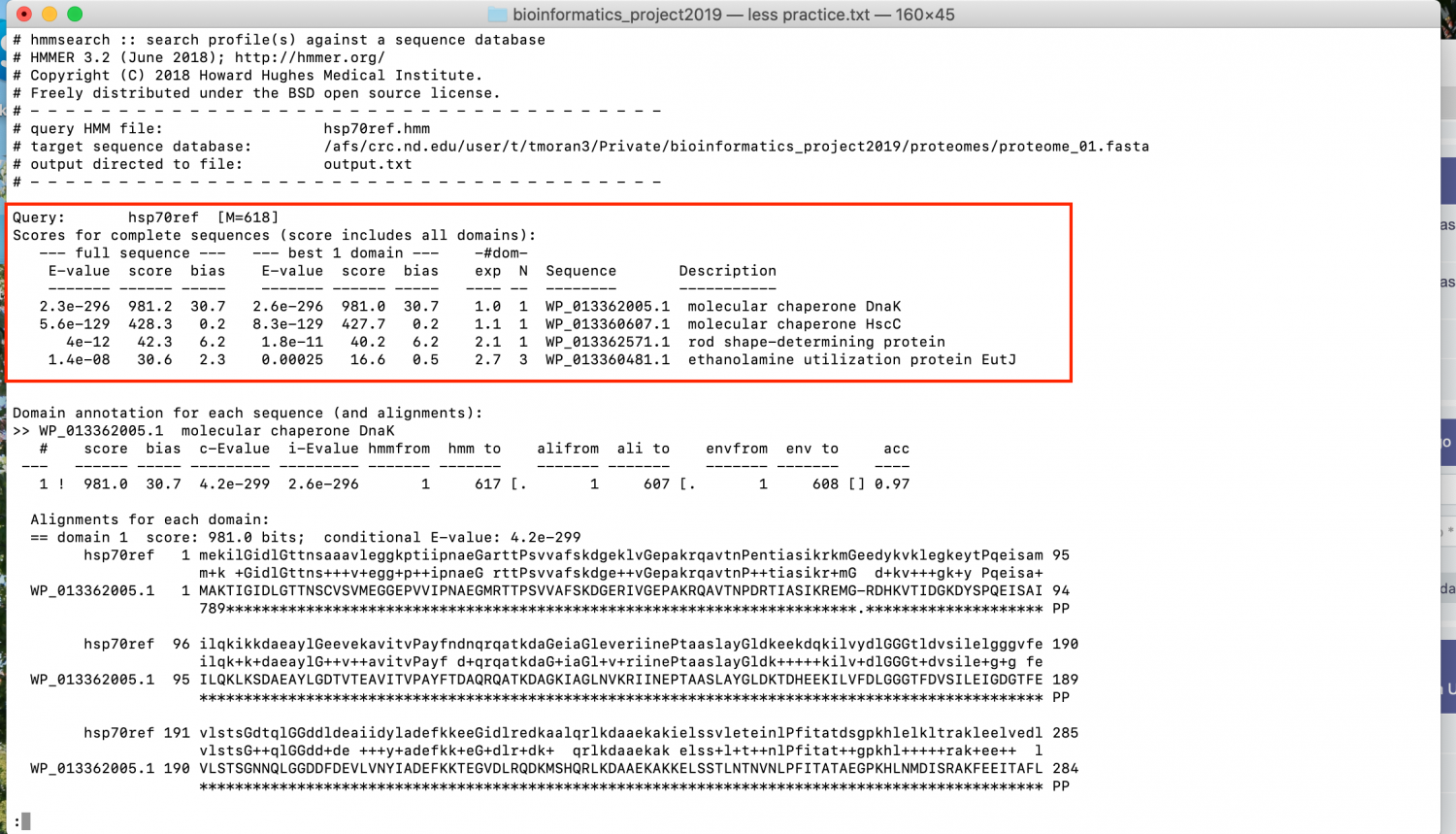
como lo puedo hacer?
Les adjunto una foto para que vean lo que quiero decir. enmarcada en rojo está la tabla que necesito copiar y pasarla a otro archivo.

Valora esta pregunta


0

